Lego.
Madmaxista
- Desde
- 28 Abr 2019
- Mensajes
- 7.364
- Reputación
- 42.324
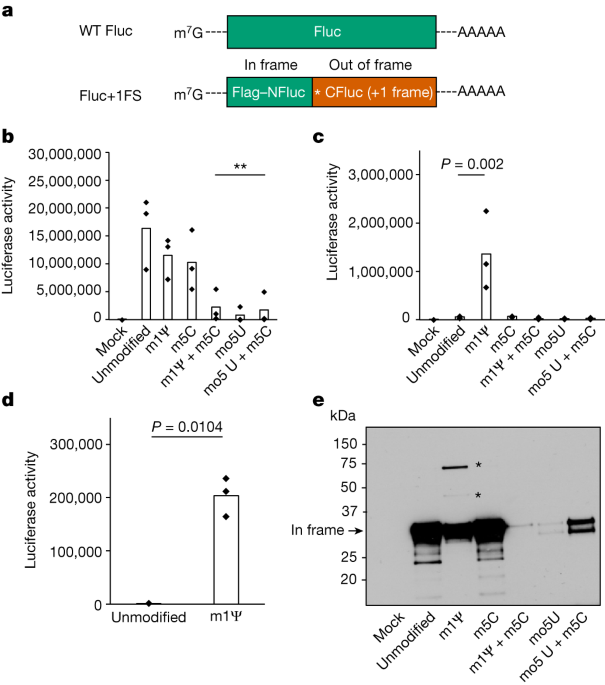
N1-methylpseudouridylation of mRNA causes +1 ribosomal frameshifting - Nature
A study demonstrates that nucleotide modifications in mRNA-based therapeutics can lead to +1 ribosomal frameshifting during translation, yielding products that can trigger immune responses.
Muy resumido: El estudio demuestra que la base artificial que han usado para el mRNA provoca que en un 10% de casos haya una mala transcripción aleatoria de la secuenciade nucleótidos del ARN, y se generan proteínas defectuosas.
Por supuesto, "no hay evidencias" de que esto sea la causa de las repentinitis ni de los muchos y diferentes efectos secundarios del pinchazo y además en la mayoría de los casos el error, la existencia de esas proteínas anómalas, no tiene consecuencias graves. (Que suena como decir "yo no digo nada pero ahí lo dejo)
El artículo es muy técnico y denso. Dejo traduccion maquineta de las conclusiones.
+1 puede producirse tras la banderillación con ARNm que contenga 1-metilΨ. Hasta donde sabemos, este es el primer informe que indica que la modificación del ARNm afecta al desplazamiento del marco ribosómico.
Además de este impacto en la inmunidad de las células T del huésped, los efectos no deseados del cambio de marco ribosómico podrían incluir una mayor producción de nuevos antígenos de células B.
Otras estrategias de modificación ribonucleotídica, como la incorporación de 5-metoxiU, disminuyeron significativamente la eficiencia de traducción de los ARNm de IVT, lo que puede limitar la traducción clínica. Aunque hemos demostrado que la traducción del ARNm N1-metilpseudouridilado conduce al desplazamiento del marco ribosómico +1 in vitro y en células cultivadas, es concebible que otros eventos de traducción errónea (como el escaneo con fugas) también podrían contribuir a las respuestas de las células T a los antígenos peptídicos con desplazamiento del marco +1.
Demostramos que los ARNm de IVT contienen pocas inserciones y deleciones de nucleótidos, y esto no cambia por la incorporación de 1-metilΨ. Nuestros datos demuestran que el desplazamiento del marco ribosómico +1 se produce en dos secuencias deslizantes caracterizadas. Por lo tanto, creemos que la banda menor de aproximadamente 50 kDa producida por la traducción del ARNm Fluc+1FS es probablemente consecuencia de varios eventos de frameshifting (Fig. 1e).
La traducción de ARNm que contiene 1-metilΨ conduce a una elongación más lenta de la traducción, causada por la alteración de la unión aminoacil-ARNt, lo que demuestra por qué no se produce el desplazamiento del marco ribosómico +1 durante la traducción de ARNm no modificado: tanto el estancamiento del ribosoma como las secuencias de deslizamiento del ribosoma parecen ser necesarios para el desplazamiento productivo del marco ribosómico +1.
Nuestros datos mecanísticos están respaldados por observaciones previas de desplazamiento de marcos ribosómicos durante la traducción de ARNm naturales, que implican el estancamiento de los ribosomas y requieren secuencias deslizantes para el desplazamiento de marcos +1.
Traducción realizada con la versión gratuita del traductor DeepL.com
Además, ZH menciona este artículo de una inmunóloga que explica un poco el paper de Nature:

That Substack about N1-methylpseudouridines and frameshifting
Another predictable harm...
Es tremendo. Traduzco:
"Todos los daños de los productos inyectables el bichito-19 eran previsibles y evitables".
Jessica Rose, PhD
Tras la última publicación de Nature titulada: "N1-methylpseudouridylation of mRNA causes +1 ribosomal frameshifting", publicada el 6 de diciembre de 20231, llegó un comentario de David Speicher, David Wiseman, Kevin McKernan, Maria Gutschi y mío titulado: "Ribosomal frameshifting and misreading of mRNA in el bichito-19 vaccines produces "off-target "proteins and immune responses eliciting safety concerns: Comment on UK study by Mulroney et al". Pudimos dar esta respuesta muy rápidamente porque ya habíamos previsto colectivamente este problema.
Los autores escriben que la N1-metilpseudouridina afecta a la fidelidad de la traducción del ARNm a través de la paralización del ribosoma, lo que induce el desplazamiento del marco. Este desplazamiento da lugar a la producción de proteínas múltiples, únicas y potencialmente aberrantes.
Los ARNm modificados para su uso en los productos el bichito-19 se optimizaron mediante codones para obtener la máxima expresión proteica en humanos. La optimización de codones, o sustitución sinónima de codones, se basa en la idea de que se pueden inducir mutaciones en un gen de interés (como la espiga) basándose en el sesgo de uso de codones de un organismo (como los humanos), para aumentar la eficiencia traslacional y la expresión proteica sin alterar la secuencia de la proteína.2 Sin embargo, es bien sabido que la optimización de codones puede dar lugar a problemas de conformación, plegamiento y estabilidad de las proteínas.
La optimización de codones podría afectar a la conformación, el plegamiento y la estabilidad de las proteínas, cambiar los sitios de modificación postraduccional e incluso afectar a su función. Las diferentes velocidades de traducción de los distintos ARNt, incluidos los que presentan un emparejamiento de bases tambaleante (un ARNt que puede reconocer varios codones sinónimos), pueden ser fundamentales para determinar la velocidad de traducción. El ribosoma puede ralentizarse y hacer una pausa durante la elongación, lo que en realidad puede ser necesario para el correcto plegamiento de la proteína. Por lo tanto, la optimización de codones puede alterar la sincronización de la traducción y, en última instancia, la función de la proteína.34
La optimización de codones también puede provocar un mal plegamiento de los ARNm debido al aumento del contenido de Guanina/Citosina (GC). Lea el preprint de McKernan et al., el artículo de Xia et al. y el artículo de Seneff et al. para obtener más información sobre los posibles problemas relacionados con la optimización de codones y los cambios en el contenido de GC. Este último grupo escribe
La sustitución sinónima de codones también provoca un cambio en los papeles reguladores y estructurales multifuncionales de las proteínas resultantes.56
De hecho, hay un enriquecimiento significativo del contenido de GC (17% y 25% de enriquecimiento según Pfizer y Moderna, respectivamente, en comparación con el SARS-CoV-2) como resultado de la optimización de codones que se hizo, y esto puede conducir a "la desregulación del sistema de unión G4-ARN-proteína y una amplia gama de posibles patologías celulares asociadas a la enfermedad, incluyendo la supresión de la inmunidad innata, la neurodegeneración y la transformación maligna "7 . El aumento del contenido de GC también altera significativamente la estructura secundaria del ARNm89 , lo que también puede provocar una pausa o un estancamiento ribosómico.
En un artículo de Nature publicado en 2011 y titulado "Breaking the silence "10 , la autora habla del peligro potencial de jugar con los codones en las proteínas terapéuticas, ya que "podría tener efectos impredecibles en la salud de las personas". Señala un estudio en el que los autores muestran que un cambio de codón sinónimo encontrado en la forma más común de fibrosis quística provoca un mal plegamiento del ARNm11. (También señala que, en el contexto del gen de resistencia a múltiples fármacos 1 (MDR1) (el gen que codifica la glicoproteína P), un cambio de codón puede interferir con las pausas que caracterizan el paso del ARN por el ribosoma, cambiando así la forma en que se pliega la cadena de aminoácidos en crecimiento.12
Pero quizá la afirmación más oportuna y espinosamente relevante de este artículo se encuentra al final, y cito:
Por el momento, las empresas que desarrollan terapias recombinantes deben verificar que la secuencia de ADN diseñada por sus científicos es la que produce sus proteínas, pero no están obligadas a indicar en qué medida difiere del código genético nativo. "No tenemos ninguna orientación sobre la secuencia [de ADN]", señala Kimchi-Sarfaty. Ese es uno de los datos que podría rastrear el sistema que ella propone. Este conocimiento, a su vez, podría ayudar a definir mejores estrategias de optimización y, posiblemente, incluso hacer que los fármacos biológicos sean más seguros para las personas.
Alla Katsnelson
Me pregunto si la FDA ha seguido alguna vez su consejo de hacer un seguimiento de las diferencias entre codones y los posibles efectos adversos resultantes.
Además de nuestro comentario sobre el artículo de Nature, un artículo de la Universidad de Cambridge titulado: "Researchers redesign future mRNA therapeutics to prevent potentially harmful immune responses" Deja claro que la conclusión más relevante del artículo de Nature es que podemos fabricar más productos igual de insanamente peligrosos que los que se inyectan en miles de millones de cuerpos porque simplemente podemos "reducir la producción de productos con desplazamiento de cuadro" mediante la "selección sinónima de secuencias resbaladizas". Por supuesto. Ahora que sabemos que las células de miles de millones de personas pueden estar produciendo proteínas aberrantes durante periodos de tiempo desconocidos, podemos simplemente esconder a estas personas bajo la alfombra, "arreglar" el producto y seguir ganando dinero.
Vamos a deslizarnos por la resbaladiza pendiente secuencial de la terapia génica directamente a las puertas del infierno.
Los fabricantes podrían haber pensado en explorar opciones para prevenir respuestas potencialmente dañinas de sus productos antes de inyectar a miles de millones de personas con ellos. Es criminal que estos productos se sigan imponiendo a recién nacidos y niños por mandato, hasta el día de hoy.
En cuanto a meterse con la progenitora Naturaleza, quizá nadie lo haya dicho mejor que Allan Drummond, biólogo celular evolutivo de la Universidad de Chicago: "Por favor, no jueguen con estos sitios; están optimizados por alguna razón", en referencia al sesgo de los codones en los mamíferos.
Después de una introducción tan larga, espero que aún tengas aguante para seguir leyendo. Mi objetivo en este artículo es facilitar a cualquier persona la explicación de esta última confirmación en el artículo de Nature: que las bases modificadas en los productos de ARNm de el bichito-19 dan lugar a un desplazamiento del marco ribosómico, y que esto puede, y probablemente lo hace, conducir a una producción aberrante de proteínas y a un mal plegamiento de las mismas.13
Tómate un café o una botella de vino y sígueme.
Figure 1: The schematic for designing an mRNA vaccine. mRNA molecules are synthesized in vitro with cap1 structure (m7GpppNm), the substitution of uridine with pseudouridine, and the use of the preferred codon in humans, optimized untranslated regions (UTR) and polyA tail sequence. These modifications result in the increase of RNA stability and translation efficiency as well as the reduction of immunogenicity. Source: International Journal of Biological Sciences 17: 1446 image No. 001.
A partir de aquí el artículo se pone muy técnico y son un motón de páginas con ilustraciones. El que lo quiera leer lo tiene en el enlace.
Mientras, seguimos metiendo esta miércoles a todo lo que se mueve, niños incluídos.
Por ejemplo...
https://www.zerohedge.com/el bichito-19/brazil-mandate-el bichito-19-shots-kids-young-6-months
Y no va a pasar nada. Da igual los ciento de miles de perecidos y millones de afectados de diversa gravedad. Y da igual en la Europa de la transparecia y la libertad que aún no se haya conseguido que se revelen las negociaciones secretas de Von der Charen con Pfizer. No va a pasar nada.
Última edición:






